SafeDrug: Dual Molecular Graph Encoders for Safe Drug Recommendations
团队介绍
论文来自伊利诺伊大学厄巴纳-香槟分校 智能医疗研究组 团队老师 Jimeng Ssun
团队研究方向:药物发现,临床试验优化,计算表型,临床预测模型,治疗建议,健康监测。
论文发表在IJCAI 2021
摘要:药物推荐是人工智能在医疗领域的一项重要任务。现有工作局限性: ①在推荐过程中没有使用一些重要的数据,例如药物分子结构。 ②药物相互作用 (DDI) 是隐式建模的,这可能导致次优结果。本文优势:①配备了全局消息传递神经网络 (MPNN) 模块和局部二分学习模块,可对药物分子的连通性和功能进行全面编码。②设计可控损失函数,可有效控制推荐药物组合中的DDI水平。
引言
longitudinal electronic health records (EHR)
丰富的电子病历数据为医疗数据挖掘提供了可能性
推荐有效和安全的药物组合是一项重要的任务,特别是对于帮助患有复杂健康状况的患者
推荐药物组合的首要目标是根据患者的健康状况为特定患者定制一种安全的药物组合
相关工作局限性
1 Inadequate Medication Encoding
每一种药物都被认为是一个(二值)单位,忽略了药物在其分子图中编码的重要药物特性,如疗效和安全性。此外,分子的亚结构与功能相关。这些知识可以用于提高用药建议的准确性和安全性
2 Implicit and Non-controllable DDI Modeling
一些现有的研究通过软约束或间接约束对药物-药物相互作用(DDI)进行建模,导致最终推荐率不可控或推荐准确度不佳
本文贡献
1 Dual Molecular Encoders
提出了双分子编码来建模药物表示,包括global and local molecule patterns
2 DDI Controllable Loss Function.
DDI约束损失,当DDI过高时进行反向优化,降低DDI率
3 Comprehensive Evaluations
实验结果提升很大,2.88% in Jaccard similarity, and 2.14% in F1 measure.
更快的训练速度,更少的参数。14% reduction in training time and around 2× speed-up during inference
论文模型整体结构
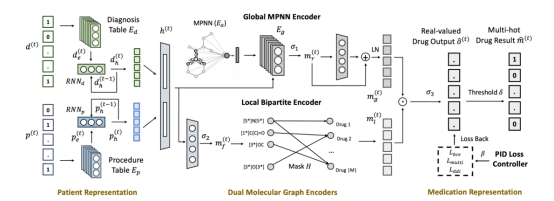
模型主要包含三个组件,Patient Representation,Dual Molecular Graph Encoder 和 medication Representation
病人表示模块首先对诊断信息、处置信息进行编码得到病人的表示向量。双分子图编码组件分布从global 和local view来学习药物中分子的表示。最后药物表示利用Liner 层进行预测。
Patient Representation

Diagnosis Embedding
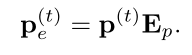
Procedure Embedding
病人表示


对于病人每次前往医院的诊断、处置信息,按照时间序列排序分别传给RNN模型进行学习。以最后时刻的RNN输出作为诊断、处置的表示。对于病人的representation。利用Liner层对二者进行拼接并映射。作为病人的的表示h。
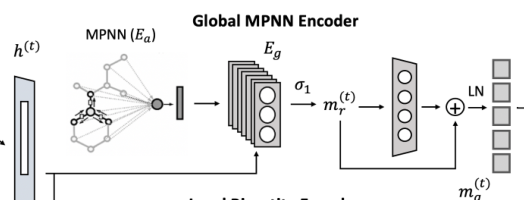
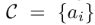 Global molecular encoder
Global molecular encoder
 首先收集出现在药物中的所有原子 ,之后设计一个可学习的编码矩阵
首先收集出现在药物中的所有原子 ,之后设计一个可学习的编码矩阵
这样对于一个给定的药物分子(假设包含原子 ),),那么我们可以得到 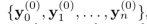 ,对应每一个原子的编码向量。
,对应每一个原子的编码向量。
采用图的消息传播机制:

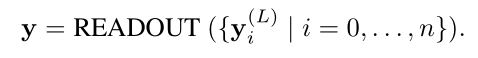
为了学习分子表示,首先对所有出现的原子进行编码。然后利用GNN进行特征聚合。
GNN聚合公式如上所示。
将每种药物都进行MPNN编码,就可以得到药物编码矩阵
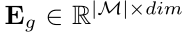
接着,计算病人与药物的匹配得分 Patient-to-drug Matching。
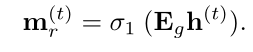
借鉴Transformer中的post-LN架构,在得分矩阵后增加前馈神经网络和层归一化
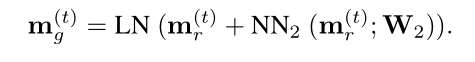
局部二部图编码 Local Bipartite Encoder:
分子分割:使用BRICS分子分割算法,在不破坏药物分子顺序的情况下,将药物分子分割成一个个官能团。另外文中给出了其他几种可行的分割方式(RECAP,ECFP)。
构建一个mask矩阵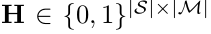 ,其中
,其中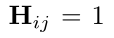 代表了第j个药物中包含了第i个官能团
代表了第j个药物中包含了第i个官能团
进行患者层面的编码 维度变换,期望结果的每一个值代表当前患者的抗病功能组合。
维度变换,期望结果的每一个值代表当前患者的抗病功能组合。
执行网络修剪,设计一个掩码单层神经网络。
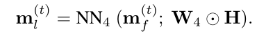
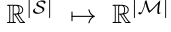
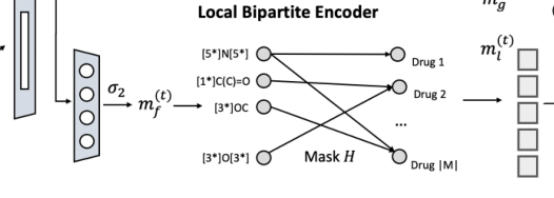
第三部分,药物表示 Medication Representation:

组件流程图如上所示,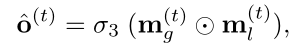
病人与药物的匹配采用的是向量元素积,过激活函数得到。
由于病人可能需要多种药物来治疗,因此药物推荐是采用的是多分类损失BCE 损失函数如下
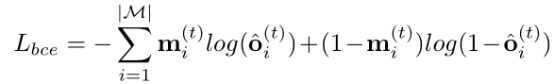
进一步的, 为了提高鲁棒性,保证真确预测的药物与错误预测的药物之间距离较远,增加了multi loss
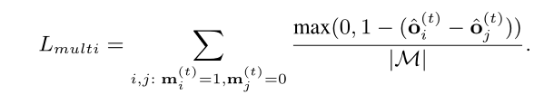
为了降低药物之间的DDI,增加了DDI loss如下
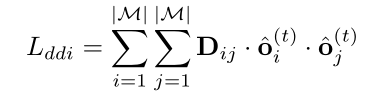
最终,对上面三个损失函数进行融合,利用超参数进行控制,保证预测准确性和较低的DDI

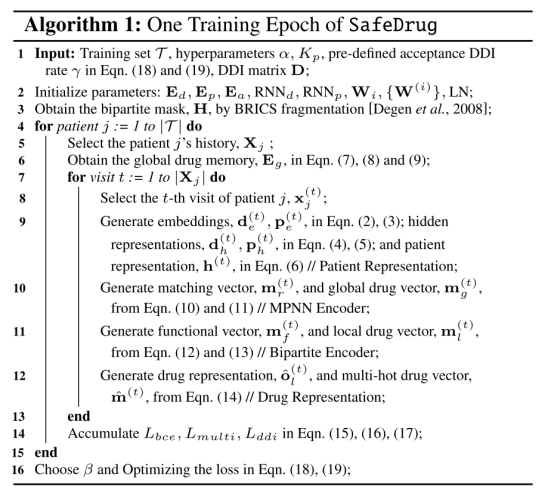
整理论文的算法逻辑伪代码如下所示
实验数据集
实验数据集选择的是MIMIC-III
MIMIC-III是一个大型的免费数据库,其中包含了与大型三级护理医院重症监护病房收治的患者的有关信息。数据包括患者生命体征、使用药物、手术代码、诊断代码、住院时间等等信息。
数据总共包括26个不同的表格,其中本次实验主要用到的表格如下:
表名 |
简介 |
包含属性 |
记录数 |
大小 |
DIAGNOSES_ICD.csv |
患者诊断ICD_9编码 |
患者编号、病案号、ICD编码 |
651047 |
4.49M |
PROCEDURE_ICD.csv |
患者手术记录ICD_9编码 |
患者编号、病案号、ICD编码 |
240095 |
1.71M |
PRESCRIPTIONS.csv |
患者用药记录 |
患者编号、药物名等等 |
4156450 |
98.6M |
实验结果如下

实验结果表明性能超过了现有的SOTA模型
模型复杂度如下
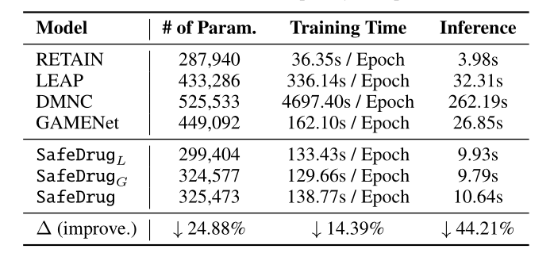
模型复杂度表示,提出了模型具有更少的模型参数,更快的训练速度。