Structure-based protein function prediction using
graph convolutional networks
1. 引言
蛋白质可以折叠成三维结构,在细胞内执行各种各样的功能。高效低成本的测序技术的出现和计算方法的进步(如基因预测)导致可用序列数量的大量增长,但人工注释的序列在数据集中占极小的比例。近几年,为了缩小序列和功能之间的差距,许多自动预测蛋白质功能的计算学方法被开发出。但在大多数情况下,对于新测序的蛋白质或研究不足的生物体的蛋白质来说,这些特征很难被发现,因为信息有限(例如,没有文本特征或生物网络可用)。在本文中,作者提出一种基于图卷积神经网络(GCNs)的功能注释蛋白质和检测蛋白质功能区域的方法,称为深度功能残基识别(DeepFRI)。该模型在公共数据集PDB和SWISS-MODEL上,展示了方法的使用性和高效性。它的数据、源代码和模型可在 beta.deepfri.flatironinstitue.org 上获得。
2. 模型
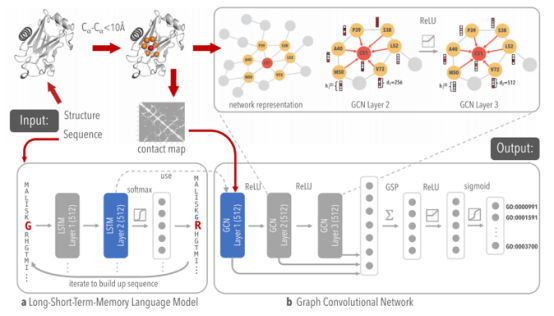
图1 论文提出的模型框架
论文提出的模型框架包含两个部分(如图1所示):
1. 序列嵌入:自监督语言模型(LSTM-LM)对蛋白质家族数据库(Pfam)中的一组蛋白质结构域序列进行预训练,用于从PDB序列中提取残基水平的特征(图1a)
2. 标签预测:在图卷积神经网络中使用深度体系结构在结构近端的残基之间传播残基水平的特征,并构建最终的蛋白质水平的特征表示(图1b)。
在GCNs的训练过程中,LSTM-LM的参数是固定的;即,LSTM-LM阶段仅作为序列特征提取器使用。为序列构建的残差级特征,与接触图一起,被用作方法的第二阶段的输入。GCNs蛋白表示是通过将GCNs所有层的特征连接到一个特征矩阵中获得的,然后被输入两个全连接层,生成所有项的最终蛋白质功能预测。模型的优点是,DeepFRI优于只考虑接触图结合简单的基线模型,表明与简化的残差特征表示相比,LSTM-LM特征显著提高了预测能力。
3. 实验部分
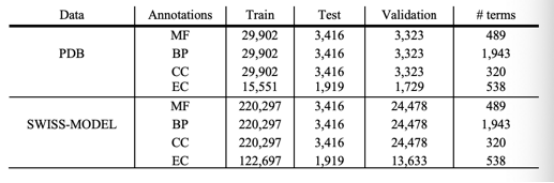
3.1数据集
表1 相关数据集统计
3.2结果展示
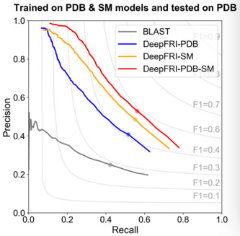
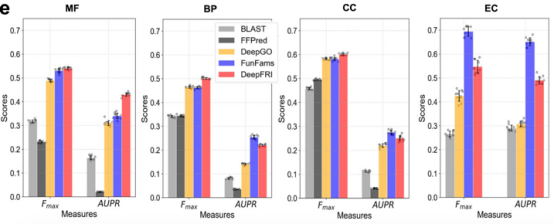
图2 模型在两个数据集上根据不同范式(GO本体和EC本体,GO本体包含MF,BP和CC分支)的预测表现
作者对于PDB和SWISS-MODEL中的每个注释链,提取其序列并构建其接触图。通过探索在PDB结构上训练的DeepFRI如何容忍建模误差,通过比较其在从SWISS-MODEL中获得的模型上和其他新结构预测协议获得的模型的性能,同时提取了大约700个实验注释的PDB链的序列(PDB700)。即使考虑到预测接触图中的误差,DeepFRI的性能(来自DMPFold和Rosetta的原生结构和模型的分别为657/0.633/0.619) 优于DeepGO方法(=0.525) (图2)
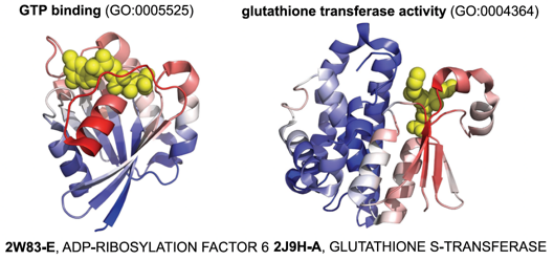
图3功能预测到蛋白质结构上的自动定位
DeepFRI通过识别与准确的GO项预测(用于MF-GO项训练的DeepFRI模型)或EC预测相关的残基来检测功能特定的结构位点。在图3中它首先计算模型的每个图卷积特征图(在MF-GO数据集上训练)对GO项预测的贡献,然后将具有积极贡献的特征图相加,获得最终的残差级激活图。
4. 总结
本文描述了将蛋白质结构与功能的预测方法,模型通过自监督和图卷积神经网络的组合,将深度学习与越来越多的可用序列联系起来,结构数据有潜力且满足越来越多的基因组序列数据所带来的注释挑战。同时,该方法为不断扩大的生命树分子视图中解释蛋白质生物多样性提供了新的见解。
参考文献
[1] Selvaraju, R. R. et al. Grad-cam: visual explanations from deep networks via gradient-based localization. ICCV,2017.
[2] Senior, A. W. et al. Improved protein structure prediction using potentials from deep learning. Nature,2020.
[3] Torng, W. & Altman, R. B. High precision protein functional site detection using 3D convolutional neural networks. Bioinformatics,2018.
[4] Kulmanov, M., Khan, M. A. & Hoehndorf, R. DeepGO: predicting protein functions from sequence and interactions using a deep ontology-aware classifier. Bioinformatics,2017.